一文读懂五大基因组技术:ChIP- ATAC-CUT&Tag-DAP-HiCuT全对比
测序技术那么多,ChIP-seq、ATAC-seq、CUT&Tag、DAP-seq、Hi-CuT相似度那么高,真是不好区分,怎么办?
别着急,且认真读完此篇汇总信息,让您简单明了的了解这五大基因组技术的区别。
Tips:文末更有特价促销信息,不要错过呦~~
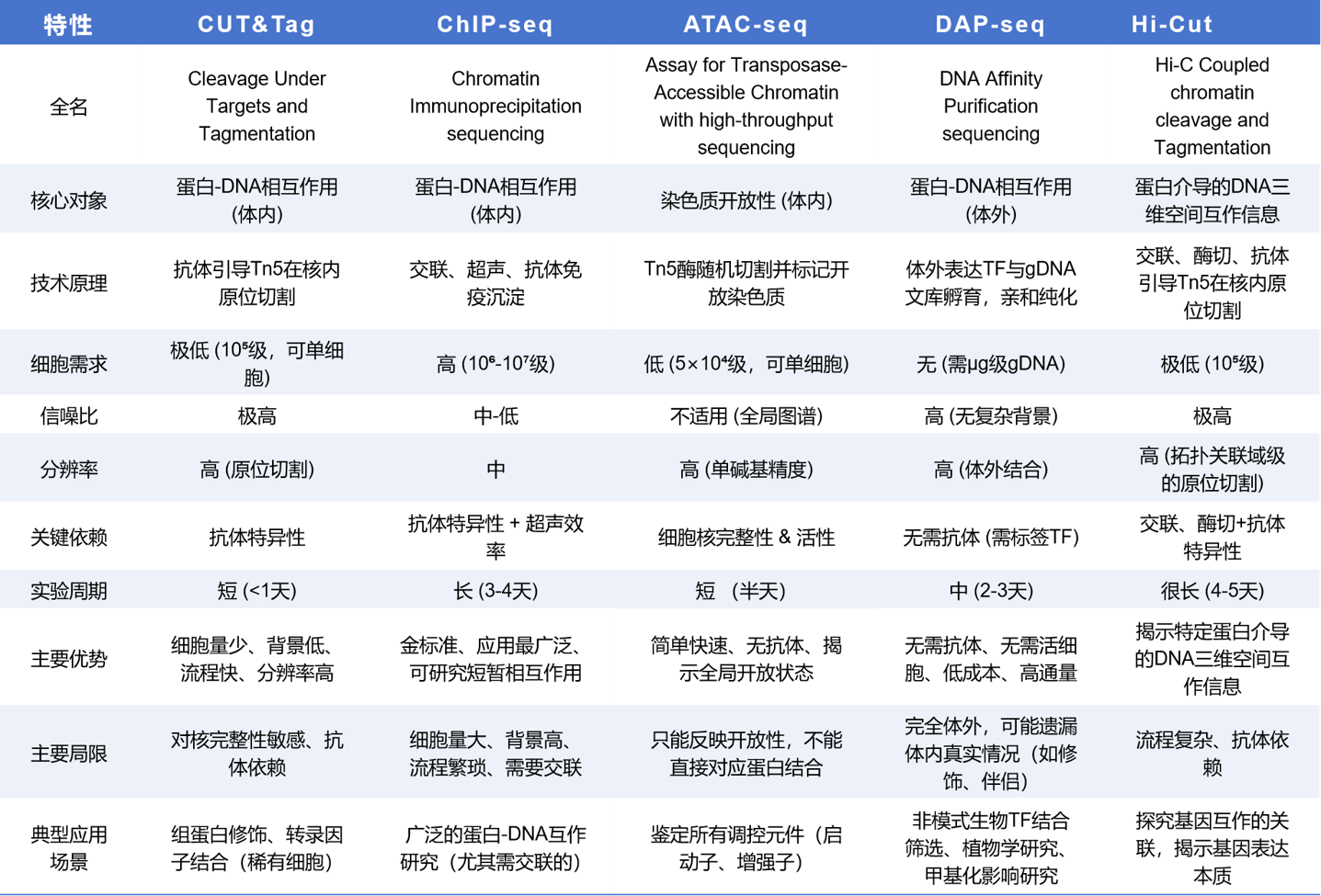
ChIP-seq (染色质免疫沉淀测序)
一种经典的、在体内(in vivo) 环境下,全基因组水平上分析蛋白质与DNA相互作用的技术。其核心是通过甲醛交联将细胞内的蛋白质与DNA固定在一起,随机断裂染色质后,利用目标蛋白的特异性抗体进行免疫沉淀,富集与该蛋白结合的DNA片段,最终通过高通量测序确定这些结合位点在基因组上的精确分布。
ATAC-seq (转座酶可及染色质测序)
一种用于直接检测全基因组范围内染色质开放性的技术。它利用一种工程化的Tn5转座酶,该酶能够高效地切割开放的染色质区域并同时连接上测序接头。开放的染色质通常代表了调控活跃的区域,如启动子、增强子和绝缘子等。因此,ATAC-seq的输出是一张全基因组的“开放染色质图谱”。
CUT&Tag (靶下切割与标签化)
一种新一代的、在体内研究蛋白-DNA互作的技术。它与ChIP-seq的目的相同,但方法学上截然不同。CUT&Tag在完整的细胞核内进行,利用特异性抗体将携带Tn5转座酶的蛋白A(pA-Tn5)融合蛋白“招募”到目标蛋白的结合位点。随后激活Tn5,使其在目标位点附近原位切割DNA并加上测序接头。这种方法避免了交联和超声破碎,因此具有背景噪音极低、信噪比极高和所需细胞量少的巨大优势。
DAP-seq (DNA亲和纯化测序)
一种在体外(in vitro) 研究DNA与蛋白质(主要是转录因子)相互作用的技术。其核心是体外表达带有亲和标签(如HaloTag)的转录因子蛋白,并将其与经片段化并加了接头的基因组DNA文库在试管中混合孵育。随后通过亲和纯化(如磁珠)回收与转录因子结合的DNA片段,进行测序。它的最大特点是完全不依赖活细胞、不需要特异性抗体,特别适用于植物和非模式生物的大规模转录因子结合位点筛选。
Hi-Cut (Hi-C Coupled chromatin cleavage and Tagmentation)
HiCuT(Hi-C Coupled chromatin cleavage and Tagmentation)是将Hi-C 3.0策略和CUT&Tag联合用于获得蛋白介导的DNA空间互作信息的创新技术,首先参考Hi-C 3.0策略,对样本进行双交联、双酶切、补平和连接,之后则按照CUT&Tag 步骤进行后续实验,包括ConAbeads 结合细胞、孵育一抗、二抗、pA/G-Tn5融合蛋白、转座、解交联、回收DNA并扩增,构建完成HiCuT文库,操作简单,效果稳定,适用于动物、活细胞等多种类样本的Hi-CUT建库过程。
二、五大基因组技术对比一览表
三、技术应用与选择指南
案例一、如想研究体内蛋白和DNA的相互作用(如转录因子结合、组蛋白修饰)
首选 CUT&Tag:如果你细胞量少、追求高信噪比和快速流程,且拥有高质量抗体。
选择 ChIP-seq:如果你的蛋白结合不稳定需要交联固定,或者CUT&Tag抗体效果不佳时,它仍是可靠的“金标准”。
选择 DAP-seq:如果你研究的是植物或难以获取大量细胞/进行遗传操作的物种,想快速、低成本地筛选多个转录因子的结合位点,且不要求体内环境。
案例二、如想全局性地看看基因组的哪些区域是“开放”的、有活性的(找增强子、启动子)
首选 ATAC-seq:这是最直接、最快速、所需细胞量最少的方法。它无需任何抗体或先验知识,就能绘制全基因组的染色质可及性图谱。
案例三、如想研究蛋白-核酸三维结构,特别是某个特定蛋白(如CTCF)介导的DNA与DNA的相互作用
选择 Hi-Cut:它在Hi-C的基础上增加了cut&tag步骤,能高效地揭示由特定蛋白介导的DNA与DNA的相互作用,是研究基因调控机制的有力工具。
案例四、样本量极少(如临床稀有样本)
优先考虑 CUT&Tag 或 ATAC-seq,它们对细胞量的要求最低。
案例五、没有好的抗体
研究蛋白结合时,可以考虑 DAP-seq(体外)。
研究染色质开放性时,ATAC-seq 是完美选择。
促销活动
sales promotion
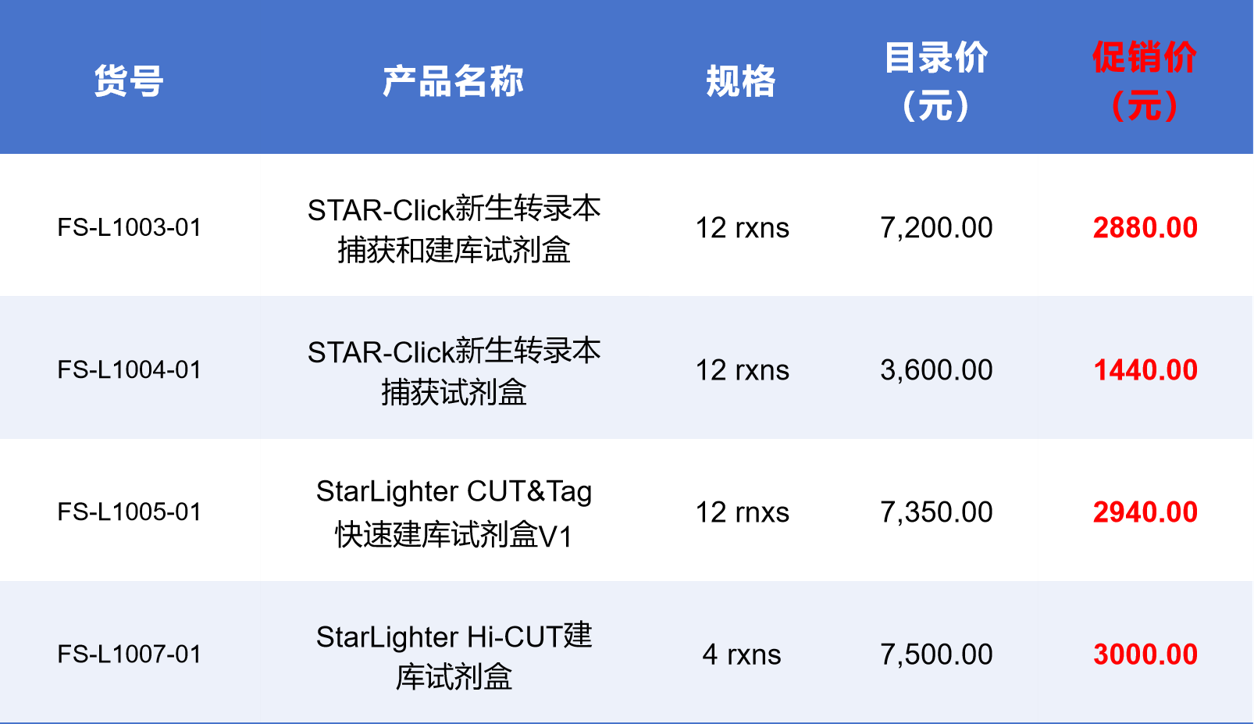
活动日期:2025.9.10-2025.10.10
扫码关注
技术与产品
新生转录本丨捕获与建库
CuT&Tag丨产品与服务



